FK-KMK UGM. Automated Sanger sequencing dianggap sebagai teknologi generasi pertama, metode yang lebih baru disebut sebagai Next-Generation Sequencing (NGS), sedangkan strategi heterogen menggunakan kombinasi persiapan template, pengurutan dan pencitraan, serta penyelarasan genom dan metode perakitan.
Perkembangan dalam teknologi sequencing ini disampaikan oleh Brendan C. Dickson, BA, BSc, MD, MSc, FCAP, FRCPC dari Mount Sinai Hospital Toronto, California dalam kegiatan Visiting Professor dan Guest Lecture yang diselenggarakan oleh Departemen Patologi Anatomi FK–KMK UGM pada Jumat (11/11). Dirinya membawakan topik diskusi “Next-Generation Sequencing in the Management of Sarcoma”.
Menurut Brendan, ada beberapa proses dasar dalam praktek NGS, yaitu ekstraksi, persiapan DNA/cDNA, pengikatan adaptor ke fragmen DNA, amplifikasi, serta analisis data. “Peran patologis dalam proses ini adalah mengidentifikasi penggunaannya, kontribusi pengambilan keputusan di dalam tim, validasi, implementasi, dan asuransi kualitas,” jelasnya.
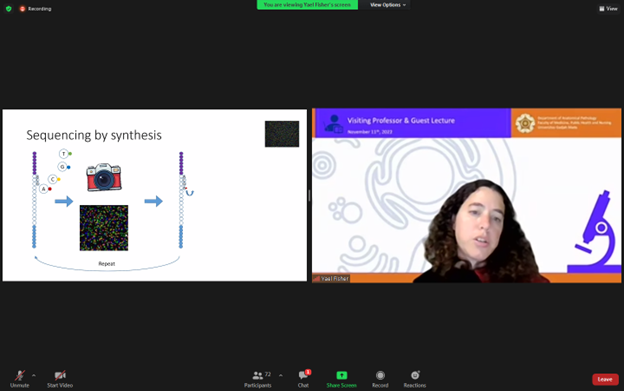
Topik diskusi kedua yang berjudul “Next-Generation Sequencing: Putting Practice into Theory” dibawakan oleh Dr. Yael Fisher, MD dari Mount Sinai Hospital Toronto, California.
Yael menjelaskan bahwa NGS library adalah kumpulan fragmen DNA dalam ukuran yang serupa. “Saat menjalankan beberapa sampel pada proses yang sama, setiap sampel ditandai dengan urutan kode batang yang unik,” jelasnya.
Dalam webinar ini juga dipaparkan mengenai perbedaan NGS amplicon based dan hybrid capture. Amplicon based lebih cocok digunakan untuk panel yang lebih kecil, memiliki harga lebih terjangkau, dan workflow lebih mudah. Sedangkan hybrid capture lebih cocok digunakan untuk panel yang lebih besar, harganya lebih mahal, serta memiliki workflow yang lebih sulit. (Nirwana/Reporter)